Science:AI模拟了5亿年的生物进化,创造了一种「史无前例」的蛋白质

蛋白质是生物中非常重要的功能分子,经过数十亿年的自然选择和演变。在这个过程中,蛋白质的序列和结构经历了无数次随机突变,并且通过生物系统的选择机制挑选,这样就形成了那些东西蛋白质具有特定的生物功能。
近几年来,伴随着深度学习和语言模型(LM)随着科学家们的发展,他们开始将这些工具应用于了解生物系统,尤其是蛋白质。
今天,Science 该杂志发表了一项重要的研究成果,展示了如何?利用语言模型生成和推理蛋白序列、结构和功能,并且提出了一个名字ESM3多模态生成模型。这种模型不仅能产生功能性蛋白质,还能产生功能性蛋白质。模拟超出 5 十亿年进化过程,产生不同于大自然已知蛋白序列的全新蛋白。

ESM3 人工智能创业公司模型由人工智能创业Evolutionary Scale研发,旨在帮助科学家理解、构思和创造蛋白质。通过这项工作,科研人员 ESM3 一种新的绿色荧光蛋白设计(GFP),它的基因序列与已知的荧光蛋白有很大的不同。一般来说,天然荧光蛋白的生物进化需要超越。 5 大约一亿年的时间。
这意味着语言模型不仅可以解读自然进化中积累的生物数据,还可以通过分析产生新的生物分子,开辟蛋白质设计和药物开发的新路径。
AI 生物语言的解码
本质上,生物是可编程的。
因为大自然中的每一个生物都共享着相同的遗传密码,而形成生命物质基础的蛋白质只是由于 20 由种植氨基酸组成。因此,有些人把它比作生命的“字母表”。
生物中复杂的蛋白质信息包含着深刻的生物规律和进化历史。近年来,科学家们通过测序基因组序列和蛋白质结构,积累了大量的蛋白质数据,包括数十亿条序列和数亿条结构信息。
随着 AI 随着科技的发展,科学家们开始使用大语言模型等深度学习模型。(LLM),“解码”这些遗传信息,通过这些模式推断和设计新的蛋白质结构和功能,揭示蛋白质序列中隐藏的深层方法和逻辑。
目前已经有多种语言模型(例如 ProtBERT、ProtGPT)它证明了蛋白质序列中的方法可以被语言模型“解码”,从而有助于理解它的功能。该领域的研究还表明,随着模型规模的扩大,语言模型的能力和准确性也有所提高。
所以,研究人员使用的超过31.5 一亿个蛋白质序列、2.36 蛋白质结构亿次,以及5.39 一亿次蛋白质数据中含有功能注解。来训练 ESM3 模型。这个模型有三种不同的规模, 14 亿、70 亿和 980 亿参数。
实验表明,随着模型参数规模的增加,ESM3 生成能力和表示学习性能都有显著提高,特别是在蛋白质结构的生成过程中,980 一亿参数模型显示出超越当前模型的强大能力。
ESM3作为该领域的前沿成果, 不只是一种传统的序列生成模式,而是一种多模式生成模式,蛋白序列、三维结构和功能同时处理。
ESM3 同时也展示了它在各种生成任务中的卓越表现。ESM3 使用了一种名为“生成掩码语言模型“方法,在输入过程中随机隐藏蛋白质的序列、结构和功能,然后通过模型推理生成缺失的部分。
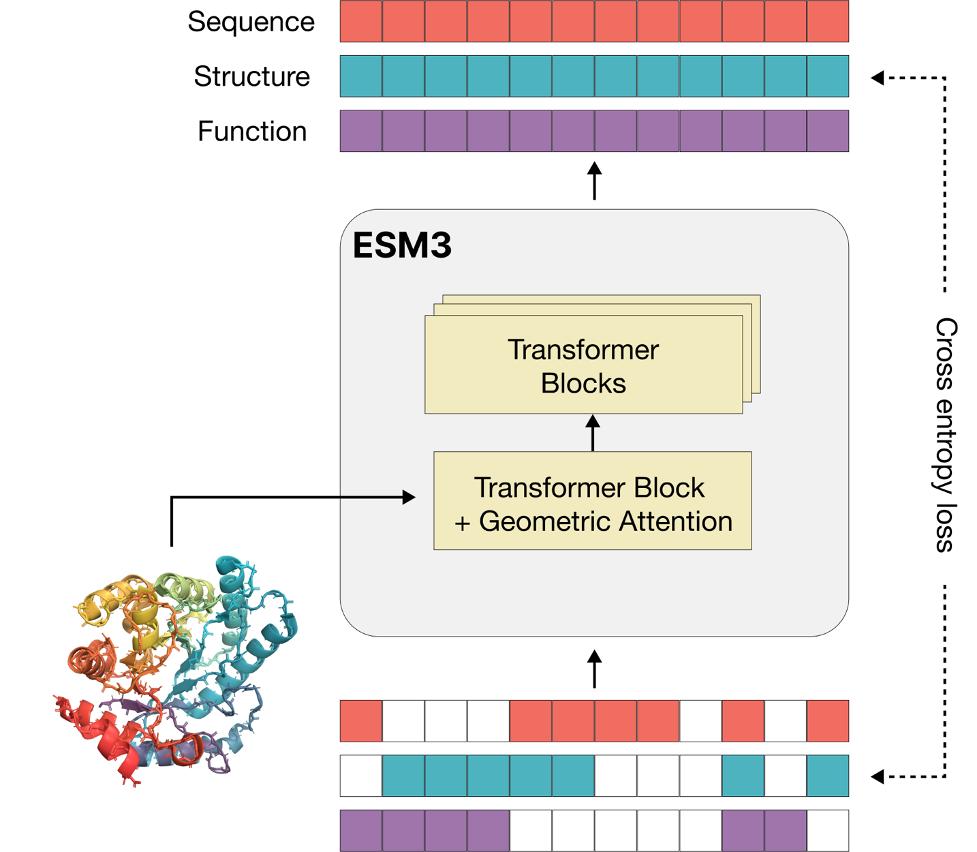
(来源:Evolutionary Scale)
研究人员通过随机掩码生成序列和结构,对比生成结果与真实蛋白质的匹配情况,发现模型能产生高质量的蛋白质序列和结构,它与真实结构的平均差异仅仅是 0.5Å。
另外,研究发现,ESM3 具有目标功能的蛋白质可以通过不同的提醒生成,这给蛋白质设计带来了极大的灵活性。ESM33不同于传统的三维空间复杂建模方法, 将三维结构分散为 token,这样,它就可以与序列和功能信息一起被输入到模型中。该方法避免了复杂的三维空间扩散结构,使生成过程更加高效和可控。
生成需 5 荧光蛋白亿年进化
为了展现了 ESM3 研究人员试图选择绿色荧光蛋白来挑战模型产生全新蛋白质的巨大潜力。
在生物学研究中,绿色荧光蛋白是一种很重要的工具,用来标记和跟踪细胞中的分子和结构。。但现有的荧光蛋白大多来自大自然,其突变一般局限于现有序列周围,很难大幅度改变其序列。在少数前提下,利润⽤⾼通量试验与机器学习,科学家只能引导。⼊至多 40-50 个突变(即 80% 同源序列),同时保留蛋白质的莹光功能。
(来源:Evolutionary Scale)
为突破这个瓶颈,研究人员通过 ESM3 特定功能提醒模型,试着生成一种全新的绿色荧光蛋白,这种蛋白质的序列与已知的绿色荧光蛋白序列相似度较低,但仍然需要保持其莹光特性。
第一,研究人员定义了一个 229 一个长氨基酸蛋白序列包含与绿色荧光蛋白有关的关键氨基酸,研究人员还提供绿色荧光蛋白的三维信息,特别是与形成莹光色素的活性位点相关的氨基酸残基。
ESM3 收到这些提醒后,模型会产生蛋白质的三维结构,尤其是氨基酸位置,以保证活性位置的良好协调。然后,在结构的基础上,模型进一步推断生成合适的氨基酸序列,并尝试保持活性位置的正确结构。
这是一个过程,ESM3 不仅仅是根据现有的绿色荧光蛋白结构生成新的序列,还可以在“已知”结构的基础上创新,生成新的蛋白质,具有低序列相似性。
经过一系列的生成和优化步骤,研究人员获得了许多新的绿色荧光蛋白,其中一种特殊的设计被称为 esmGFP。这个新的蛋白质和现有的荧光蛋白质(例如 tagRFP)序列之间的相似度为 58%,与最接近的天然蛋白(eqFP578)的序列不同 107 个体氨基酸,序列相似度为 53%。
研究人员还进一步验证了绿色荧光蛋白是否具有实际的莹光功能。结果表明,虽然 esmGFP 亮度特征延迟,改善时间长,但最终的亮度与已知的绿色荧光蛋白相似,具有稳定的亮度特征。。
研究人员还提供了时间校准系统的发展分析,指出通过当前蛋白质的自然进化过程获得 esmGFP,就需要超出 5 等效时间为亿年。
ESM3 未来的潜力和应用
ESM3 另外一个显著的亮点是它在多模态条件下的产生和控制。
换句话说,研究者可以通过提醒特定的蛋白质结构、功能或关键氨基酸来生成新的蛋白质,以满足这些条件。。例如,模型可以在保持整体结构完整性的同时生成具有特定功能位点的蛋白质。
此外,模型还可以通过组合不同的提示生成符合复杂要求的蛋白质。例如,研究人员提醒蛋白质的二次结构和功能关键词,并生成与这些提醒高度一致的蛋白质。
ESM3 该模型具有提醒响应能力和可控性,促使其在蛋白质设计领域具有很高的实用价值,尤其是在生成与现有已知蛋白质有显著差异的新型蛋白质方面。
在 ESM3 在模型的帮助下,研究人员不仅可以制作出新的绿色荧光蛋白,而且可以在设计上进行创新,突破自然进化的局限。这为蛋白质工程、合成生物学和药物开发等未来领域提供了新的概率,也为蛋白质设计和功能验证提供了更高效的工具。
例如,ESM3与自然进化相比。 在基础研究和应用研究方面,蛋白质设计的速度可以大大加快,产生在自然界中无法轻易获得的新蛋白质,这是一个巨大的突破。
另外,在药品设计领域,通过生成具有特定功能的蛋白质是一项重要的研究内容, 研究人员可以制造出符合特定目标的蛋白质,ESM3,减少实验验证的时间和成本。
但是在合成生物学领域,ESM3 有助于开发新的生成方法,生成具有新功能的酶或代谢方法。
研究人员还指出,随着模型规模和信息量的进一步增加,ESM3 蛋白质具有更复杂、更创新的潜力。。ESM3在未来 的应用可能包括从基础研究到药物设计等更多领域,为蛋白工程开辟全新概率。
现在,ESM3 已通过 API 通过编程或基于浏览器的互动式测试版,科学家可以推出公开测试版。 app 设计蛋白质。科学家可以使用免费的学术浏览层。 EvolutionaryScale Forge API,也可采用开放式模型代码和权重。
本文来自微信微信官方账号“学术头条”(ID:SciTouTiao),作者:木木,36氪经授权发布。
本文仅代表作者观点,版权归原创者所有,如需转载请在文中注明来源及作者名字。
免责声明:本文系转载编辑文章,仅作分享之用。如分享内容、图片侵犯到您的版权或非授权发布,请及时与我们联系进行审核处理或删除,您可以发送材料至邮箱:service@tojoy.com



