追问|科学家如何还原和挖掘海洋微生物基因数据?
·GOMC数据库为未来的研究提供了大量的资源,包括开发新的生物技术产品、探索微生物的生态功能、研究微生物与环境变化的关系、开发新的疾病治疗方法等。
基因是生命的“源代码”,不仅包含了自然进化的奥秘,也包含了无数的生物技术资源。如何获取、分析和应用基因数据是现代生物学的核心问题之一。
最近,一个研究小组分析并深入挖掘了目前已经公开的海洋微生物宏基因组数据,建立了迄今为止最完整的海洋微生物基因数据库,并发现了包括新基因编辑工具、抗菌肽和PET塑料降解酶在内的各种具有应用潜力的基因资源。这项研究于2024年9月4日发表在《自然》中。(Nature)杂志上,团队成员来自华大生命科学研究院、山东大学、东安格利亚大学、中国海洋大学、厦门大学、丹麦哥本哈根大学等机构。
据估计,超过90%的物种还没有被发现,海洋覆盖了地球表面约71%的区域。细菌、古菌、病毒等微生物是海洋世界中数量最多的“居民”,1毫升海水约100万个,是海洋生态的重要组成部分。
从海水中取样并对其中的遗传信息进行测序,得到的所有基因都是“宏基因组”,没有区分。(Metagenomics)。对研究人员来说,从这些庞大的基因数据中获取有用的信息是一个巨大的挑战。
在这项研究中,研究团队持续了五年,通过近240年的公开研究, 重新分析Tb海洋微生物宏基因组信息,构建了海洋微生物组数据库Thee,拥有超过4.31万个海洋微生物基因组和24.58亿个基因序列。 Global Ocean Microbiome Catalogue(GOMC),海洋生态系统包括从南极到北极,从近海到深海,从表面海洋到万米超谷底等多样化。其中,20,000多种微生物是潜在的新发现物种,近10,000种微生物是在深海等独特生物中首次发现的。
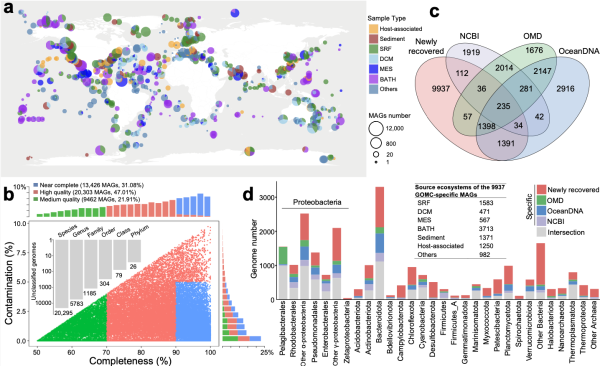
通过数据挖掘,研究小组发现了大量可用于基因编辑、抗生素和塑料降解的基因资源,如海洋微生物基因组大小变化、遗传免疫机制演变等生态规律。
科学家如何从基因碎片中恢复完整的基因序列?从基因上看,海洋生态有哪些神奇的规律?基因数据中可以找到哪些有用的医疗和环境资源?为了回答这些问题,本报科技近日采访了该探索的通信作者、中国大学生命科学研究院青岛分院院长范广益博士。
化零为整:宏基因组装技术
微生物群落非常复杂,宏基因组技术非常有效,因为大多数微生物无法在实验室中培养,获取环境样本中的所有基因信息。这项技术的难点之一是如何从这些混合的基因信息中恢复单个物种的基因组。
范广益告诉澎湃科技,该研究采用宏基因组装分箱技术,拼接和分箱聚类环境样本测序获得的大量基因短序列,从而获得完整的基因组。这些基因组被称为“宏基因组装基因组”(Metagenome-assembled genomes, MAGs)。
宏基因组装涉及大量的比较和计算。它是DNA或RNA大分子中的一个核苷酸序列。经过测序仪的测序,科学家可以得到一个序列的“碎片”,被称为“读者”(reads)。通过对比这些“碎片”之间的重叠部分,我们可以将它们连接起来,获得更长的连续序列,这就是所谓的“重叠群”(contigs)。
接下来,科学家需要通过重叠群序列中显示的方式来判断这些序列是否为基因组,这些序列是否为基因组。这个过程被生动地称为“分箱”。(binning)。与MAG相对应的是,相同的序列被归入一个“盒子”。
近年来,随着高通量测序技术的发展,宏基因组数据迅速增加,组装技术成为探索新类别和基因数据的有力工具。范广益提到,在这项研究中,研究团队整合了大量已经公开的海洋微生物宏基因组数据库,涵盖了从极地到赤道、从海洋表面到深海的广阔海洋环境,极大地拓展了对海洋微生物多样性的认识。
虽然宏基因组装技术带来了许多新的发现,但其产品的生物真实性仍然存在争议。MAGs的真实性不仅受到样品被其他物质污染的影响,而且由于难以验证而受到质疑,尤其是在相应物种尚未被发现的前提下。
对于这一点,范广益认为,随着测序技术的进步和生物信息分析方法的完善,MAGs的准确性和生物真实性得到了显著提高。研究小组通过物种系统的发展分析,对宏基因组数据进行了质量控制,提高了分箱的准确性,并对其进行了分箱分析。通过这种方式获得的MAGs可以代表海洋微生物的真实基因组,为了了解微生物群落的结构和功能提供重要信息。
基因组大小及免疫分布:基因视角下的海洋微生物群落
这项研究发现了一些有趣而重要的海洋微生物生态规律,通过分析重构的基因组数据。
在生物世界中,生物的复杂性与基因组的大小无关。例如,无恒变形虫是一种单细胞原生动物,其基因组由6700亿对碱基组成,而人类只有30亿对。什么因素影响基因组的大小和功能是一个重要的问题。
范广益表示,基因组大小是微生物适应环境变化的重要指标之一,与微生物的代谢复杂性、生存策略和进化历史密切相关。研究表明,大基因组细菌通常存在于自然条件复杂、资源丰富的海洋环境中。
研究人员推测,这些细菌的基因组会包含更多的基因,给它们更多样化的代谢方式和生理功能,从而适应多变的海洋环境。然而,大型基因组也给细菌复制和维护带来了挑战。
除基因大小外,研究还发现,海洋微生物免疫系统中不同免疫策略的分布存在微妙的平衡关系。
和人类一样,一些海洋微生物,如细菌和古菌,也受到病毒的威胁,抗生素可以杀死细菌,这也是他们的“宿敌”。对于病毒,很多微生物都有一个CRISPR-Cas系统,其中CRISPR是一个重复的基因,它可以包含和识别曾经攻击过细菌的病毒DNA并摧毁它,而Cas是一种负责裁剪和摧毁与这些基因相关的蛋白质。而且面临着“有毒”的抗生素,微生物已经演变成抗生素抗性基因(ARGs),赋予微生物对一种或多种抗生素的抵抗力。
研究表明,在不同的生态环境中,微生物在高温下倾向于有更多的CRISPR-Cas系统,如热液喷嘴(海底的特殊地质结构)等。同时,这两种策略的分布似乎是相互平衡的。当微生物中的CRISPR-Cas系统增加时,ARGs的总数就会减少,它们在整体分布中会保持一定的平衡。
范广益提到,这种平衡可能反映了微生物在维持其遗传稳定性和适应环境压力(如抗生素选择压力)方面的复杂相互作用。CRISPR-在一定程度上,Cas系统可以限制ARGs的水平传播,但是它可以保护微生物免受外源核酸入侵。此外,CRISPR-Cas系统本身也可能受到许多因素的影响,如微生物基因结构、自然条件、宿主-病原菌相互作用等。
“这一发现强调,在分析微生物遗传系统如何适应和抵御环境挑战时,应考虑各种因素和复杂的生态动态。这也为进一步研究微生物抗性基因的传播机制、开发新的抗菌策略和保护微生物资源提供了重要的科学论证。”他说。
基因编辑、抗生素、塑料分解:挖掘基因“宝藏”
生命的“中心”是基因:DNA携带的遗传信息通过RNA转录和翻译合成蛋白质,最终形成各种组织结构来实现生命功能。基因包含了无数资源,可以应用于医疗、环境和工业。在这项研究中,研究人员发现了许多“宝藏”基因,通过挖掘GOMC数据库。
研究小组首先识别了多个新的CRISPR-Cas9系统。科学家们发现,该系统可用于基因编辑,在药物开发、基因治疗等领域具有很大的价值,并在2020年获得了诺贝尔化学奖,这是一种可以切割外源入侵DNA的免疫机制。
范广益提到,这次发现的新型CRISPR-Cas9系统具有不同的识别特异性、靶向效率和编辑精度,有助于对现有的基因编辑工具进行优化和定制,以提高特定生物系统中的编辑效率和精度。另外,在特殊环境下,海洋微生物的适应性可能会给CRISPR-Cas系统带来独特的稳定性和活性,这对基因编辑工具的开发可以在特定环境下工作非常重要。
这项研究还通过生物合成基因簇聚基因进行(BGCs,一组基因在基因组中相邻并参与化合物生成)进行预测,确定了多种抗菌肽(AMPs)。抗菌肽是一种小分子肽,它们通过破坏细菌细胞膜或影响细胞中的关键生物过程来抑制或杀死微生物。范广益表示,GOMC鉴定的新抗菌肽在序列和结构上与已知的抗菌肽有显著差异,这表明它们可能对特定微生物人群有独特的功能机制或活性。
随着抗生素在人类中的广泛应用,细菌的耐药性逐渐成为威胁,新型抗生素亟待开发。范广益告诉《ThePaper》,新发现的抗生素呈现出对各种细菌的抑制作用,包括一些具有抗性的传统抗生素,为开发新的广谱抗生素提供了可能。
这项研究还发现了多种对PET塑料有显著活性的水解酶。PET塑料全称“聚对苯二甲酸乙二醇酯”,是应用最广泛的塑料材料之一,常用于饮料瓶、食品包装。PET水解酶是一种能够催化PET塑料降解的生物催化剂,能够对PET塑料中的酯键水解进行特定的识别和催化,将塑料分解成较小的分子,从而启动塑料降解过程,对解决全球塑料污染问题具有重要意义。
范广益表示,这些从数据库中发现的水解酶表现出嗜盐和热稳定性,这些独特的生长习惯很可能是他们在深海等恶劣生活中生存和发挥活力的重要因素。这也意味着他们在面对工业领域常见的苛刻条件时,仍然可能保持高效的催化活性。
“我们详细分析了筛选出来的PETases的生化特性。资料表明,他们在特定条件下对PET薄膜的降解效率很高。例如,研究中提到的dsPETase05水解酶可以在3天内降解大部分PET膜,降解率达到83%,比已知的IsPETase活性高44倍。”范广益说。
“GOMC数据库为未来的研究提供了大量的资源,包括开发新的生物技术产品、探索微生物的生态功能、研究微生物与环境变化的关系、开发新的疾病治疗方法等。”范广益说。
本文仅代表作者观点,版权归原创者所有,如需转载请在文中注明来源及作者名字。
免责声明:本文系转载编辑文章,仅作分享之用。如分享内容、图片侵犯到您的版权或非授权发布,请及时与我们联系进行审核处理或删除,您可以发送材料至邮箱:service@tojoy.com





