AlphaFold 3不开源,统一生物语言大模型阿里云先开源。
在大模型中放入169861个生物物种数据,大模型实际上获得了生物中心法则的奥秘。——
不仅可以识别DNA、RNA与相应蛋白质的关系,SOTA模型也可以与基因分类、蛋白质相互作用预测、热稳定性预测等7种不同类型的任务相媲美。
模型名叫LucaOne,由阿里云飞天实验室生物智能计算团队打造。
与AlphaFold相比 3由于没有开源,650多名学者联合指责LucaOne训练推理代码及相关数据目前都是已开源。
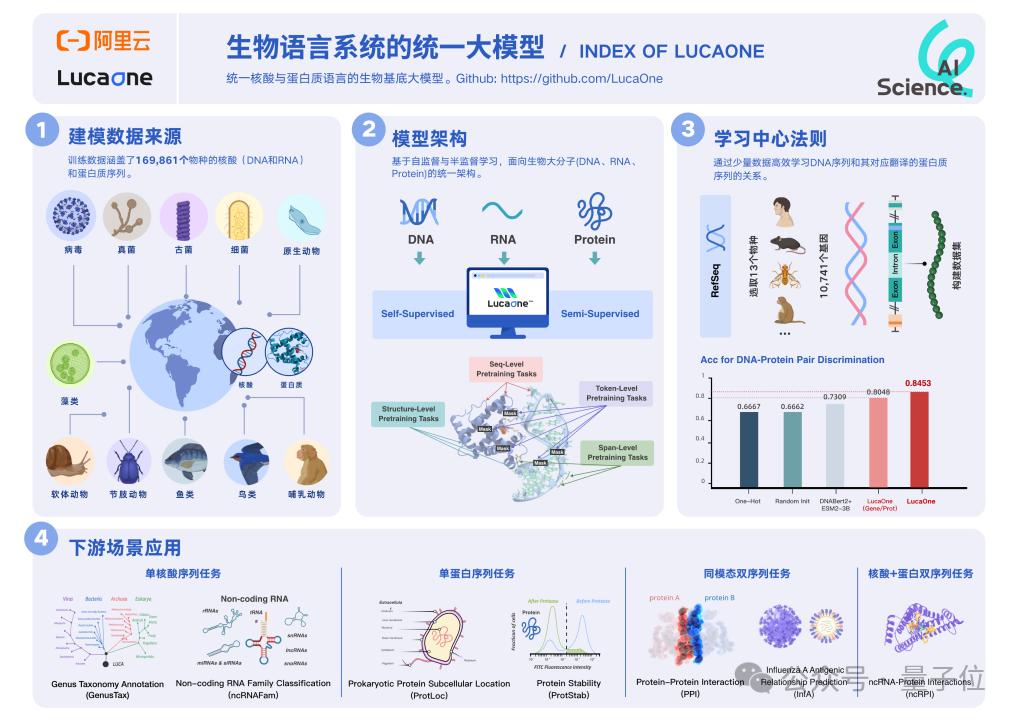
目前,LucaOne是第一个核酸语言全生物系统 蛋白质语言与底座模型相结合。也就是说,LucaOne由核酸组成(DNA、RNA)与蛋白质序列联合训练而来。
研究人员通过一系列实验发现,它可以广泛地应用于各种下游任务。
核酸序列及其对应蛋白的正负样本数据集中在13个物种中,关系总数为24000,LucaOne提供预测准确率0.85的表征模型。。
ESM-3B组合远高于目前行业内最好的预训练模型。 单核酸训练版本的DNAbert2(0.73)和其它建模方法也明显高于LucaOne。 单个蛋白质训练版。
预测流感H3N2病毒疫苗的有效性(免疫逃逸风险)等其它任务,LucaOne精确率可以达到100%。
同时,量子位也与论文一作有关,谈到了LucaOne的实现细节,以及AI。 for 生物科学领域Science的发展。
联合训练核酸和蛋白质序列
一般来说,LucaOne围绕中心规则的数据构建,可以学习中心规则背后的原理和逻辑,提取蛋白质翻译过程中固有的复杂方法和关系,相当于使用中的基因转录和提供一对DNA、 RNA、没有区别的蛋白质表征。
PS:
生物学的中心法则是遗传信息从DNA传递给RNA,然后从RNA传递给蛋白质的过程。这个过程包括DNA的拷贝、RNA的转录和蛋白质的翻译。
从这个角度来看,LucaOne的整个工作流程就是这样的阿姨:
从技术上讲,构建LucaOne的难点首先是构建数据集。
真正存在于生命科学领域的只是分子数据。
例如,表示核酸的方法有四种碱基。DNA是腺嘌呤。(A)、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T);腺嘌呤是RNA(A)、鸟嘌呤(G)、胞嘧啶(C)、尿嘧啶(U)。蛋白质由氨基酸组成,大约有20-22种氨基酸存在于自然界,每种氨基酸也用一个字母表示。
为了了解这些分子的特点和功能,人类通常需要添加大量的注释信息,包括一些图片的注释。注释信息属于人类语言,自然本身是不存在的,从“自然”语言到“人类文化”语言,在生命科学领域形成了一种跨模式。
所以LucaOne的预训练数据不仅包含DNA、RNA、三类分子的数据(核苷酸序列或氨基酸序列)是蛋白质的序列,同时也使用了这些分子。注解信息。
核酸和蛋白质序列和注解信息共计169861个物种,分为两部分:
来自RefSeq的核酸数据包括核酸序列和注释;来自InterPro、UniProt、ColabFold、RCSB-PDB、AlphaFold2,包括蛋白质序列、注解和三维结构。
据悉,阿里云飞天实验室与中山大学、浙江大学等多个团队合作,收集数据集。
另外一个难点是生物分子序列的预测不同于下一个token的大语言模型预测,在模型训练阶段也需要一些特殊的设计。
使用LucaOneTransformer-Encoder该结构由20个编码器块组成,嵌入层为2560,总参数为2560。1.8B。
在此基础上,研究人员进行了一些改进:
用Pre-Layer归一化取代Post-Layer归一化,从而更好地训练深层网络;
嵌入旋转位置(RoPE)为了推理更长的序列,取代传统的绝对位置代码。
另外,核苷酸和氨基酸在数据处理和模型训练过程中以统一的方式进行表征或编码。使用token-type 通过核酸和蛋白质序列的混合训练,embeddings可以区分核苷酸(0)和氨基酸(1)。
研究人员还加入了两项自监督掩码任务,八个半监督预训练任务,通过序列注解增强模型对数据的理解。
已经能够理解基因与蛋白质的关系。
为了验证核酸和蛋白质数据混合训练的优势,研究人员利用核酸和蛋白质数据独立训练了两个额外的模型。——LucaOne-Gene和LucaOne-Prot,并且使用相同的5.6M 在生物学中心法则任务中,checkpoint进行了比较。
使用t-SNE可视化表明,与其它模型相比,LucaOne的嵌入在两个数据中显示出更密切的聚类,可能包含更多的上下文信息。
研究人员设计了一项数据和评估任务,以验证LucaOne通过广泛学习基因和蛋白语言,具备理解生物学中心规则中基因和蛋白质之间关系的能力。
核酸序列选择13个物种及其对应蛋白质的正负样本数据,关系总数为24000,其中正负样本比例为1。:2。基因序列信息是其在基因组中的原始数据,包括许多非编码区域(包括子、控制元件、垃圾片段等)。
选择训练:验证:测试比例为::3:25;也就是只有3200组数据作为训练,18750组数据作为测试集在数据组中预测核酸序列是否可以翻译成蛋白序列。。
因此,LucaOne提供了一个表征模型。0.85预测准确率不仅远高于ESM-3BB组合ESM-3B目前行业内最好的预训练模型。 单核酸训练版本的DNAbert2(0.73)和其它建模方法也明显高于LucaOne。 单个蛋白质训练版。
这说明这两种大分子数据联合训练可以显著提高模型的学习效果。
有趣的是,研究人员在模型细分表现中发现,LucaOne海鞘这种生物的预测性能相对较差(其他模型也相似)。进一步分析了海鞘的特点,表明海鞘使用中心规则的具体规则——密码子的偏好与其他生物因进化适应性等各种原因明显不同。
他们猜测LucaOne可能会使用另一种中心法则语法。“方言”,而且这一“方言”在训练数据集中只有100条,所以模型对这一规则的学习不够好。
LucaOne在其他下游任务中也广泛应用于不同类型的下游任务。
具体而言,研究人员对7个不同类型的下游生物计算任务进行了评估,包括:
单独的序列任务:GenusTax(属分类)、ncRNAFam(ncRNA家族分类)、ProtLoc(定位蛋白质亚细胞)、ProtStab(预测蛋白质热稳定性)。
对于任务,同源序列:InfA(分析流感血凝素)、PPI(预测蛋白质相互作用)。
对于任务,异源序列:ncRPI(ncRNA-预测蛋白质相互作用)。
为了简化下游任务,研究人员采用了三种简单的网络结构,对应不同的输入方式:
结果显示,GenusTax、ProtStab、ncRNAFam、InfA、在PPI任务中,LucaOne明显优于其它模型;在ProtLoc任务中,LucaOne相当于ESM2-3B,优于SOTA。;LucaOne在ncRPI任务上优于DNABert2 搭配ESM2-3B:
值得注意的是,在预测流感H3N2病毒免疫逃逸风险时,研究人员利用1968年至2010年间分离的大规模H3N2病毒HA序列数据,进行了基于流感毒株抗原联系的预测模型。
通过病毒HA抗原序列,我们可以预测它是否会诱发HIA实验的血凝,从而预测它是否会在特定人群中发生免疫逃逸。
要知道,目前预防和控制流感最有效的方法是接种流感疫苗,但由于流感病毒的快速变异率,无法及时准确地推荐与流感病毒相匹配的流感疫苗株。根据WHO和CDC的监测,流感疫苗的有效性在40%-60%之间。因此,准确预测流行毒株,判断免疫逃逸风险是一个重要而困难的问题。
基于LucaOne的研究人员使用 一层感知器模型达到100%的精度。
它还表明,LucaOne学习了大量的核酸序列信息,其中包含了大量的其它病毒序列,为计算特定任务提供了良好的信息补充。
有兴趣的家庭成员可以查看更多细节的原文。
“Science for AI”
如前所述,LucaOne背后的开发团队来自阿里云飞天实验室LucaTeam,LucaTeam也与多个团队进行了深度合作。
中山大学医学院施莽教授及其团队参与了LucaOne模型数据的设计和验证。施莽教授认为:
LucaOne是一个极其重要的尝试。令我惊讶的是,LucaOne可以更有效地学习核酸和蛋白质之间的关系,而不需要任何先验知识。
北京协和医学院中国医学科学院病原生物研究所优势,美国微生物科学院会士舒跃龙在流感病毒方面,教授及其团队参与了LucaOne的分析与验证。舒跃龙教授说:
将前沿的AI技术与病原生物学相结合,具有重要的科学意义和社会价值。通过这种紧密的跨学科合作,我们可以探索更多的病原生物发源进化、跨种传播和传染病规律,为传染病的防治和生物安全做出更大的贡献。
另外,论文一作贺勇作为阿里云飞天实验室生物计算高级算法专家,我们还与LucaOne进行了AI聊天。 for 生物科学领域Science的发展。
在他看来,AI for Science在生物科学领域处于起步阶段。考虑到AI的可解释性,他们现在基本上只把AI当成一个工具,但现在他们正在发展AI。 for Science是一个很好的时间节点。
由于测序技术发展迅速,测序成本大大降低,分子序列数据可以轻松获取。有了大量的数据积累,数据驱动的AI就可以了。 for 与Science相关的工作。
然而,目前的局限性在于测序或分子的序列数据,而现实世界中每一个分子的存在都是一个分子。空间布局,这样做可能需要更复杂的模型来处理。
不同学科之间的研究方法不同,微观世界的探索也受到当前设备技术的制约。贺勇认为,人类对生物科学领域的理解只是冰山一角,无法从全局的角度构建一个全面通用的系统。
最后他还补充道:
现在大家都在AI上 for Science越来越重视,相当于用AI来解决具体的问题。接下来,我想我们应该回顾一下Science能给AI带来什么。处理具体问题是第一步。事实上,应该考虑反馈技术本身。
论文链接:https://www.biorxiv.org/content10.1101/2024.05.10.5927v10
GitHub链接:https://github.com/LucaOne
本文来自微信微信官方账号“量子位”(ID:QbitAI),作者:西风,36氪经授权发布。
本文仅代表作者观点,版权归原创者所有,如需转载请在文中注明来源及作者名字。
免责声明:本文系转载编辑文章,仅作分享之用。如分享内容、图片侵犯到您的版权或非授权发布,请及时与我们联系进行审核处理或删除,您可以发送材料至邮箱:service@tojoy.com


